Author: 〇東 孝太郎1,2、山口 雅也1、中田 匡宣3、武部 克希4、住友 倫子1、鈴木 守5、川端 重忠1
Affiliation: 1阪大 院歯 口腔細菌、2阪大 院歯 義歯・高齢 、3鹿大 院医歯 口腔微生物、4阪大 院歯 口外2 、5阪大 蛋白研
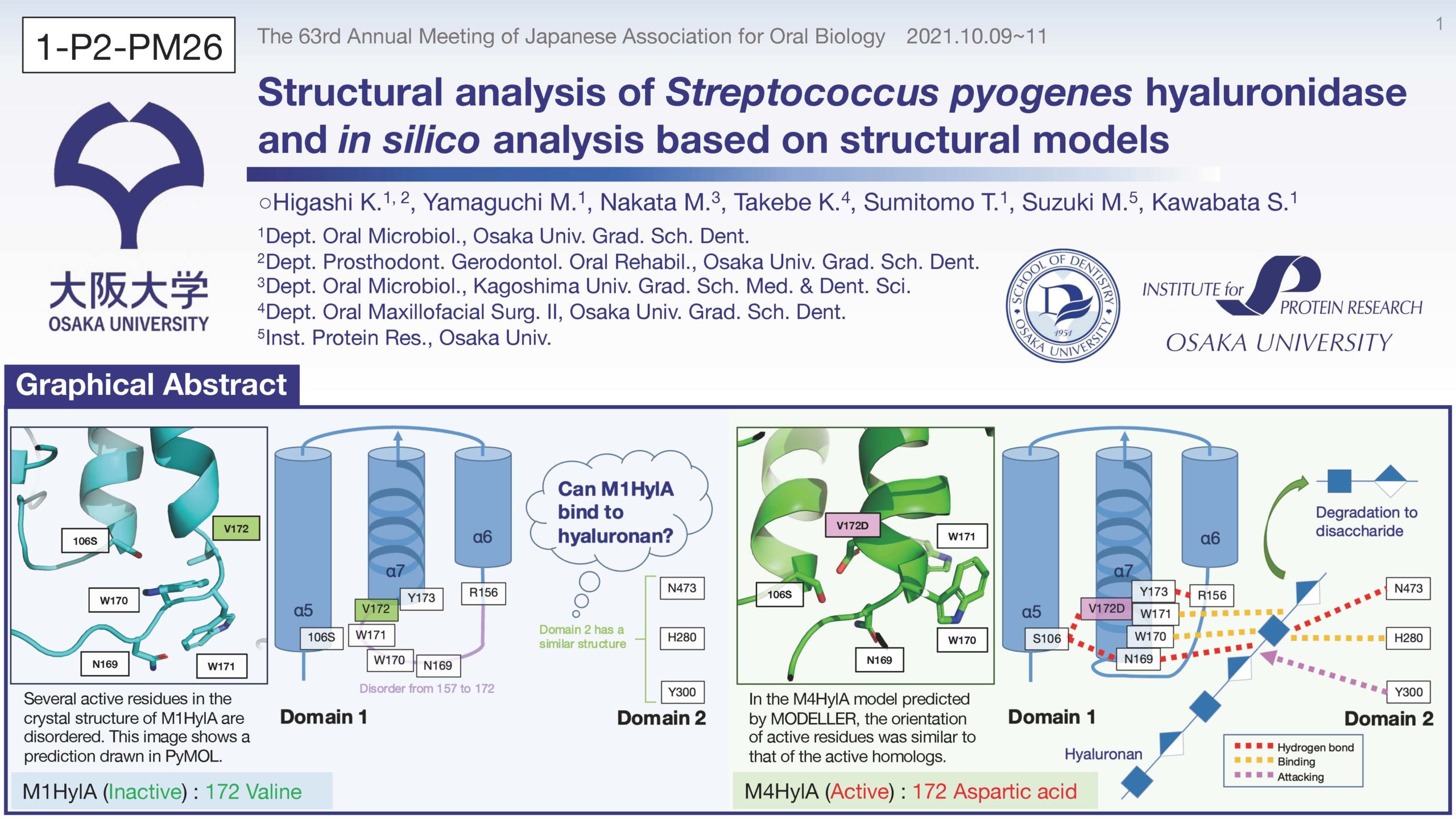
Abstract: 化膿レンサ球菌は、咽頭炎や扁桃炎などの局所性感染症の原因菌であるとともに、壊死性筋膜炎などの全身性の劇症型感染症を引き起こす。肺炎球菌やB群レンサ球菌は、病原因子であるヒアルロン酸を二糖に分解する酵素を産生する。しかし、emm4型やemm22型などを除く化膿レンサ球菌の大部分は、不活性型のヒアルロン酸分解酵素HylAを保有している。活性の有無は172番目のアミノ酸1残基の変異によって生じている。本研究では、化膿レンサ球菌のHylAについて1残基変異がタンパク質の構造、および機能に及ぼす影響を検討した。
emm1型化膿レンサ球菌の不活性型HylAについて、X線結晶構造解析にて構造を決定した。肺炎球菌ホモログの機能部位との相同残基は、ほとんどが構造的に保存されていた。不活性型HylAの172番目のバリン側鎖が水素結合を形成できず、その周辺に位置する一部の活性残基がDisorder領域となり、ヘリックスを形成せずにループとなっていた。つまり、活性残基の向きが安定しないことで酵素活性を示さないことが示唆された。一方で、ヒアルロン酸との結合能についてAutoDock vinaを用いて予測したところ、結合可能であることを示唆する複合体構造のモデルと結合エネルギーが得られた。また、構造予測プログラムMODELLERを用いて、今回決定した構造と既報の他のレンサ球菌のホモログの構造から、活性型であるemm4型のHylAの構造を予測したところ、不活性型HylAでDisorder領域となっていた172番付近の残基においてヘリックス構造が形成され、活性残基の側鎖の向きが一定となることが示唆された。
これらの結果から、活性中心外の1残基の変異により周辺の二次構造が変化し、ヒアルロン酸の分解活性は欠失したことが示唆された。一方で、結合部位を構成する残基は構造的に保存されているため、糖鎖結合能は有している可能性が示された。![]()
 メールで問い合わせ
メールで問い合わせ
[1-P2-PM26] 化膿レンサ球菌の不活性型ヒアルロン酸分解酵素の
結晶構造解析と活性型変異体の構造予測
 微生物
微生物+1


コメント